 Esta es una traducción del artículo del biólogo molecular Ian Musgrave, si bien es algo antiguo (con un par de errores) y no está actualizado a los muchos avances en el campo de la abiogénesis, consideramos que es un artículo muy valioso para aclarar los temas de las falacias estadísticas referentes a la probabilidad de la vida.
Esta es una traducción del artículo del biólogo molecular Ian Musgrave, si bien es algo antiguo (con un par de errores) y no está actualizado a los muchos avances en el campo de la abiogénesis, consideramos que es un artículo muy valioso para aclarar los temas de las falacias estadísticas referentes a la probabilidad de la vida.
Introducción
Glosario
Acriltransferasa:
Una enzima o ribosoma que sintetiza péptidos.
Ligasa:
Una enzima o ribosoma que agrega un monómero a un polímero, o enlaza dos polímeros cortos.
Monómero:
Cualquier subunidad simple de un polímero. Un aminoácido es un monómero de un péptido o proteína, un nucleótido es un monómero de un oligonucleótido o polinucleótido.
Nucleótido:
Adeína, Guanina, Citocína y Uranina. Estos son los monómeros que componen los oligo/polinucleótidos tal como el ARN.
Oligonucleótido:
Un polímero corto de subunidades de nucleótidos.
Polimerasa:
Una enzima o ribosoma que hace un polímero fuera de los monómeros. Por ejemplo, el polimerasa ARN hace al ARN salir de simples nucleótidos.
Ribosomas:
Un catalizador biológico hecho desde el ARN.
Autorreplicante:
Una molécula que puede hacer identicas o casi identicas copias de sí misma a partir de pequeñas subunidades. Se conocen al menos cuatro autorreplicantes.
Bastante a menudo alguien viene con la afirmación de que “la formación de cualquier enzima por azar es casi imposible, por lo tanto la abiogénesis es imposible”. A menudo se cita un impresionante cálculo del astrofísico Fred Hoyle, o sacan a relucir la llamada “Ley de Borel” para probar que la vida es estadisticamente imposible. Estas personas, incluido Fred, han cometido uno o más de los siguientes errores.
Problemas con los cálculos creacionistas “tan improbables”
1) Calculan la probabilidad de la formación de una proteína “moderna” o aún peor una bacteria completa con todas sus proteínas “modernas”, por eventos aleatorios. Esto no es la abiogénesis en absoluto.
2) Asumen que la vida requiere un número fijo de proteínas con sequencias fijas para cada proteína.
3) Calculan la probabilidad como intentos secuenciales en vez de intentos simultáneos.
4) Malinterpretan que es lo que se entiende por cálculo de probabilidades.
5) Subestiman seriamente el número de enzimas/ribozomas presentes en un grupo de sequencias aleatorias.
Trataré y los llevaré a través de estos errores, y les mostraré por qué no es posible hacer un cálculo de la “probabilidad de abiogénesis” de ninguna manera.
Un glóbulo protoplásmico primordial
Así que el cálculo acerca de la probabilidad de la formación de 300 proteínas de aminoácidos largos (esto es, enzimas como la carbonoxipeptidasa) dados aleatoriamente es (1/20)^300 o 1 posibilidad en 2.04 x 10^390, que es asombrosa y extremadamente improbable. Esto sumado a la probabilidad de generar 400 o más enzimas similares, se alcanza una figura tan grande que el solo hecho de contemplarla causa que tu cerebro comience a salirse por las orejas. Esto da la impresión de que la formación del más pequeño organismo parezca totalmente imposible.
Sin embargo, esto es completamente incorrecto.
Primeramente, la formación de polímeros biológicos desde monómeros es una función de las leyes de la química y la bioquímica, y estas definitivamente no son aleatorias.
En segundo lugar, toda la premisa es incorrecta desde el principio, porque en las modernas teorías del abiogénesis la primera “cosa viviente” sería mucho más simple, ni siquiera una protobacteria o una preprotobacteria (lo que Oparin llamaba un protobionte,[8] y Woese llama un protogenote[4]), sino que una o más moléculas simples probablemente no más largas que de 30 o 40 subunidades. Estas simples moléculas luego evolucionaron en sistemas cooperativos autoreplicantes, y finalmente en organismos simples.[2, 5, 10, 15, 28] La siguiente ilustración es una camparación entre un protobionte hipotético y una bacteria moderna.
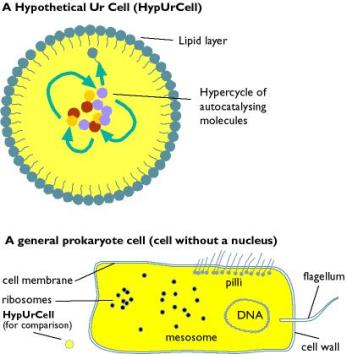
La primera “cosa viviente” podría haber sido una simple molécula autoreplicante, similar a los péptidos “autorreplicantes” del grupo de Ghadiri [7][17], o los hexanucleótidos autorreplicantes [10], o posiblemente un polímero de ARN que actúa sobre sí mismo.[12]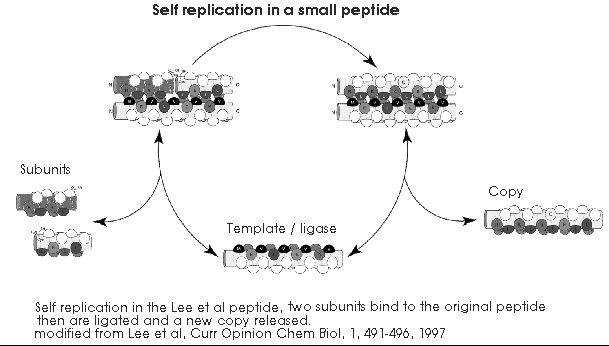
Otra visión es que los primeros autorreplicantes fueron grupos de catalizadores, ya sean enzimas proteicas o ribozomas de ARN, estos se regenan a sí mismos como un ciclo catalítico.[ 3 , 5 , 15 ,26 , 28 ] Un ejemplo es la subunidad autorreplicante SunY tres. [ 24 ] Estos ciclos catalíticos podrían estar limitados a un charco pequeño o laguna, o ser un complejo catalítico absorbido ya sea por arcilla o material lípido en la arcilla. Dado que hay muchas secuencias catalíticas en un grupo de péptidos o polinucleótidos aleatorios no es improbable que se formara un pequeño complejo catalítico.
Los dos modelos no son mutuamente excluyentes. Los peptidos Ghadiri pueden mutar y formar ciclos catalíticos.[ 9 ] No interesa si los primeros autorreplicantes fueron moléculas simples o complejo de moleculas simples, este modelo no tiene nada que ver con el tornado de Hoyle pasando por un basurero para formar un 747. Solo para establecer el punto, aquí está una comparación simple de la teoría que es criticada por los creacionistas y la actual teoría de la abiogénesis.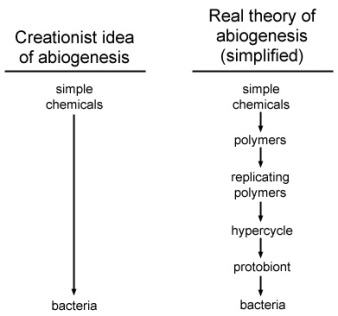
Notar que la teoría real tiene un número de pequeños pasos intermédios, y, de hecho, he dejado algunos afuera (especialmente entre los estados de hiperciclos y protobiontes) para simplificar. Cada paso está asociado a un pequeño incremento en la organización y la complejidad, y los elementos químicos ascienden hacia lo orgánico en vez de dar un gran salto.[ 4 , 10 ,15 , 28 ]
De dónde los creacionistas sacaron que los modernos organismos aparecieron espontaneamente es incierto. La primera formulación moderna de la abiogénesis, la hipótesis Oparin / Haldane de los años 20, comienza con proteínas / proteinoides simples desarrollandose lentamente en células. Incluso las ideas que circulaban de 1850 no fueron teorías de “espontaneidad”. ¡Lo más lejano que se puede ir es a las ideas originales de Lamarck de 1803!.[ 8 ]
Dado que los creacionistas han estado criticando una teoría de hace 150 años que ningún biólogo moderno sostiene, ¿para qué ir más lejos? porque hay problemas fundamentales en las estadísticas y la bioquímica que aparecen en estas “refutaciones” erradas.
El mito de la “secuencia de la vida”
A veces se escucha otra pretención, que hay una “secuencia de la vida” de 400 proteínas, y que las secuencias de aminoácidos de esas proteínas no pueden ser cambiadas, para que los organismos estén vivos.
Esto, sin embargo, son disparates. La pretención de las 400 proteínas parece venir del genoma codificado de proteínas del Mycobacterium genetalium, el cual tiene el genoma más pequeño actualmente conocido para un organismo moderno.[20] Sin embargo las inspecciones del genoma sugieren que podrían estar reducidas aún más hasta un conjunto mínimo de genes de 256 proteínas.[20] Notar otra vez que esto es un organismo moderno. El primer protobionte / progenote prodría haber sido aún más pequeño[4], y estar precedido por aún más simples sistemas químicos.[3, 10, 11, 15]
En cuanto a la afirmación de que las secuencias de proteínas no se pueden cambiar, de nuevo esto no tiene sentido. Hay en la mayoría de las regiones de las proteínas donde casi cualquier aminoácido puede ser substituido, y otras regiones donde las sustituciones conservadoras pueden ser hechas (donde los aminoácidos cambiados pueden serlo con otros aminoácidos, neutrales por otros aminoácidos neutrales, aminoácidos hidrofóbicos por otros aminoácidos hidrofóbicos). Algunas moléculas funcionalmente equivalentes pueden tener diferencias entre 30 y 50 % en sus aminoácidos. De hecho es posible sustituir proteínas bacterianas no idénticas estructuralmente por proteínas de levadura y proteínas de gusanos por proteínas humanas y el organismo felizmente seguiría vivo.
La “secuencia de la vida” es un mito.
Lanzamiento de monedas para principiantes y ensamble macromolecular
Asi que juguemos al juego creacionista y miremos la formación de péptidos por suma aleatoria de aminoácidos. Ciertamente esta no es la forma en que los péptidos se formaron en la Tierra temprana pero será instructivo.
Usaré como ejemplo el péptido “autorreplicante” del grupo Ghadiri mencionado más arriba.[7] Podría usar otros ejemplos, tal como el autorreplicante hexanucleótido[10], el autorreplicante SunY[24] o el polimerasa ARN descripto por el grupo Eckland[12], pero para continuar con la historia de las pretenciones creacionistas un péptido pequeño es lo ideal. Este péptido tiene un largo de 32 aminoácidos con la secuencia de RMKQLEEKVYELLSKVACLEYEVARLKKVGE y es una enzima, un ligasa péptido que hace una copia de sí mismo de dos subunidades de 16 aminoácidos de longitud. Es además del tamaño y composición ideal para ser formado por síntesis abiótica de péptidos. El hecho de que es un autorreplicante es una ironía agregada.
La probabilidad de generar esto en sucesivos intentos aleatorios es (1/20)^32 o 1 en 4.29 x 10^40. Esto es mucho, mucho mas probable que 1 en 2.04 x 10390 del escenario creacionista estándar de “generar carboxypeptidasa por azar”, pero aún es absurdamente bajo.
Sin embargo, hay otro lado en estas estimaciones probabilísticas, y depende del hecho de que la mayoría de nosotros no tenemos noción de las estadísticas. Cuando alguien nos dice que algún evento tiene una chance en un millón de ocurrir, muchos de nosotros esperamos que se hagan un millon de intentos antes de que el evento ocurra. Pero esto es incorrecto.
Aquí tienes un experimento que puedes hacer tú mismo: toma una moneda, arrójala cuatro veces, escribe los resultados y hazlo de nuevo. ¿Cuántas veces piensas que tendrás que repetir este procedimiento (intento) antes de obtener 4 caras en una sola tanda?
La probabilidad de tener 4 caras en una tanda es ahora de (1/2)^4 o 1 chance en 16: ¿tenemos que hacer 16 intentos para obtener 4 caras (CCCC)? NO, en experimentos sucesivos obtuve 11, 10, 6, 16, 1, 5, y 3 intentos antes de que CCCC apareciera. La figura 1 en 16 (o 1 en un millón o 1 en 1040) da la probabilidad de un evento en un intento dado, pero no dice cuándo ocurrirá en una serie. Puedes obtener CCCC justo en el primer intento (yo lo conseguí). Aún en una chance en 4.29 x 10^40, un autorreplicante podría aparecer sorprendentemente temprano.
Pero aún hay más.
Una chance en 4.29 x 10^40 aún es asquerosamente inverosímil. Es difícil manejar este número. Aún con el argumento de arriba (podrías obtenerlo en el primer intento) la mayoría de la gente diría “seguramente tomaría más tiempo que el que la Tierra ha existido hasta obtener este replicador por métodos al azar”. No, realmente. En el ejemplo de arriba estábamos examinando intentos secuenciales (uno detrás del otro), como si hubiera una sola proteína / ADN / proto-replicador siendo ensamblado en cada intento. La verdad es que habría millones de millones de intentos simultáneos con los millones de millones de moléculas en bloque que trabajaron recíprocamente en los océanos, o en los miles de kilómetros de costas que pudieron proveer superficies catalíticas o moldes [2,15].
Volvamos a nuestro ejemplo con las monedas. Digamos que se toma un minuto en lanzar la moneda 4 veces; para generar CCCC tomará en promedio 8 minutos. Ahora consigue 16 amigos, cada uno con una moneda, para que todos lancen las suyas 4 veces simultaneamente; el promedio de tiempo para generar CCCC es ahora un minuto. Ahora traten de conseguir 6 caras en una tanda, esto tiene la probabilidad de (1/2)^6 o 1 en 64. Esto tomaría una media hora en promedio, pero salgan y consigan 64 personas, y ahora vuelven a tener 1 minuto de tiempo promedio. Si quieres obtener una secuencia con una chance de 1 en mil millones, solamente recluta a la población de China para que lancen monedas para ti y tendrás esa secuencia en prácticamente nada de tiempo.
Así que si en nuestra Tierra prebiótica tenemos mil millones de péptidos creciendo simultáneamente, eso reduce el tiempo para generar nuestro replicador muy significativamente. Ok, estas mirando ese número otra vez, una chance en 4.29 x 10^40, ese es un número Muy grande, y si bien mil millones de moléculas primordiales son muchas moléculas, ¿podríamos obtener alguna vez suficientes moléculas para ensamblar aleatoriamente nuestro primer replicador en menos de la mitad de mil millones de años?
Sí, por supuesto, un kilogramo de aminoácido tiene 2.85 x 10^24 moléculas en él (esto es más de un billón); una tonelada tiene 2.85 x 10^27 moléculas. Si tomáramos un semiremolque cargado de cada uno de los aminoácidos y los tiramos en el medio de un lago tendrías suficientes moléculas como para generar nuestro replicador en particular en menos de 10 años, dado que puedes hacer 55 proteínas de aminoácidos largos en una o dos semanas.[14,16]
Entonces, ¿cómo encaja esto en la Tierra prebiótica? En la Tierra temprana es probable que los océanos hallan tenido un volumen de 1 x 10^24 litros. Dada una concentración de aminoácidos de 1 x 10^-6 M (una sopa moderadamente diluida, ver Chyba y Sagan 1992) [23] hay aproximadamente 1 x 10^50 potenciales cadenas primigenias, así que un número razonable de ligasa péptidos eficientes (alrededor de 1 x 10^31) se podrían producir en casi un año, déjenlos solos un millón de años… La síntesis de autorreplicantes primitivos podría suceder relativamente rápido, aún dada la probabilidad de 1 chance en 4.29 x 10^40 (y recuerda, nuestro replicador podría ser sintetizado justo en el primer intento).
Asume que toma una semana en generar una secuencia.[14,16] Entonces el ligasa Ghadiri podría generarse en una semana, y cualquier secuencia citocroma C podría generarse en apenas un millón de años (junto con alrededor de la mitad de todas las secuencias de péptidos posibles, una gran proporción de las cuales serán proteínas funcionales de algún tipo).
Si bien he usado la ligasa Ghadhiri como un ejemplo, como mencioné más arriba los mismos cálculos pueden ser hechos para los autorreplicantes SunY, los polimerasas de ARN, de Eckland. Dejo esto como ejercicios para el lector, pero en general la conclusión es la misma para estos oligonucleótidos.
Espacios de busqueda o ¿cuántas agujas hay en un pajar?
Así, he mostrado que generar una pequeña enzima cualquiera no es extremadamente difícil como los creacionistas (y Fred Hoyle) sugieren. Otra mala interpretación es que la mayoría de la gente cree que el número de enzimas / ribosomas, dejando de lado los polimerasas de ARN o cualquier forma de autorreplicantes, representa una configuración muy inverosímil y que las chances para que una simple formación de enzimas / ribosomas, dejando de lado el número de ellos, desde una suma aleatoria de ácidos / nucleótidos es muy pequeña.
Sin embargo, un análisis hecho por Eckland sugiere que en el espacio de secuencia de 220 nucleótidos de ARN largos, 2.5 x 10^112 secuencias son ligasas eficientes.[12] No está mal para un compuesto previamente pensado para ser solo estructural. Volviendo a nuestro océano primitivo de 1 x 10^24 litros y asumiendo una concentración de nucleótidos de 1 x 10^-7 M[23], entonces hay más o menos 1 x 10^49 cadenas de potenciales nucleótidos, así que un número razonable de ligasas de ARN eficientes (alrededor de 1 x 10^34) podrían ser producidas en un año, déjenlas solas un millón de años… El potencial número de polimerasas de ARN es además, alto, alrededor de 1 en cada 10^20 secuencias es una polimerasa de ARN[12]. Consideraciones similares se aplican para las transferasa acrilribosomicas (alrededor de 1 por cada 1015 secuencias), y para las síntesis de nucleótidos ribosómicos [1, 6, 13].
Así que, aún con más números realísticos, el ensamblaje aleatorio de aminoácidos en sistemas con “soporte de vida”, parecería ser bastante factible, aún con los números más pesimistas para la concentración de monómeros originales [ 23 ] y el tiempo de síntesis.
Conclusiones
La gran premisa de los calculos de probabilidad de los creacionistas es incorrecta en primer lugar porque se basa en la teoría incorrecta. Más aún, este argumento a veces está contaminado con falacias estadísticas y biológicas.
En este momento, desde que no tenemos idea de cuán probable es la vida, es virtualmente imposible asignar probabilidades significativas a ninguno de los pasos de la vida excepto a los primeros dos (monomeros a polímeros p=1.0, formación de polímeros catalíticos p=1.0). Para los polímeros replicantes a la transición de hiperciclo la probabilidad bien puede ser 1.0 si Kauffman tiene razón acerca del “cierre catalítico” en sus modelos de transición de fase, pero esto requiere química real y modelación más detallada para confirmarlo. Para la transición hiperciclo -> protobionte, la probabilidad es dependiente de conceptos teóricos que aún están siendo desarrollados y es desconocido.
Sin embargo, al final, la factibilidad de la vida depende de la química y la bioquímica que siguen siendo estudiadas, y no lanzando monedas al aire precisamente.
Referencias
[1] Unrau PJ, and Bartel DP, RNA-catalysed nucleotide synthesis. Nature, 395: 260-3, 1998
[2] Orgel LE, Polymerization on the rocks: theoretical introduction. Orig Life Evol Biosph, 28: 227-34, 1998
[3] Otsuka J and Nozawa Y. Self-reproducing system can behave as Maxwell’s demon: theoretical illustration under prebiotic conditions. J Theor Biol, 194, 205-221, 1998
[4] Woese C, The universal ancestor. Proc Natl Acad Sci USA, 95: 6854-6859.
[5] Varetto L, Studying artificial life with a molecular automaton. J Theor Biol, 193: 257-85, 1998
[6] Wiegand TW, Janssen RC, and Eaton BE, Selection of RNA amide synthases. Chem Biol, 4: 675-83, 1997
[7] Severin K, Lee DH, Kennan AJ, and Ghadiri MR, A synthetic peptide ligase. Nature, 389: 706-9, 1997
[8] Ruse M, The origin of life, philosophical perspectives. J Theor Biol, 187: 473-482, 1997
[9] Lee DH, Severin K, Yokobayashi Y, and Ghadiri MR, Emergence of symbiosis in peptide self-replication through a hypercyclic network. Nature, 390: 591-4, 1997
[10] Lee DH, Severin K, and Ghadri MR. Autocatalytic networks: the transition from molecular self-replication to molecular ecosystems. Curr Opinion Chem Biol, 1, 491-496, 1997
[11] Di Giulio M, On the RNA world: evidence in favor of an early ribonucleopeptide world. J Mol Evol, 45: 571-8, 1997
[12] Ekland EH, and Bartel DP, RNA-catalysed RNA polymerization using nucleoside triphosphates. Nature, 383: 192, 1996
[13] Lohse PA, and Szostak JW, Ribozyme-catalysed amino-acid transfer reactions. Nature, 381: 442-4, 1996
[14] Ferris JP, Hill AR Jr, Liu R, and Orgel LE, Synthesis of long prebiotic oligomers on mineral surfaces [see comments]. Nature, 381: 59-61, 1996
[15] Lazcano A, and Miller SL, The origin and early evolution of life: prebiotic chemistry, the pre- RNA world, and time. Cell, 85: 793-8, 1996
[16] Ertem G, and Ferris JP, Synthesis of RNA oligomers on heterogeneous templates. Nature, 379: 238-40, 1996
[17] Lee DH, Granja JR, Martinez JA, Severin K, and Ghadri MR, A self-replicating peptide. Nature, 382: 525-8, 1996
[18] Joyce GF, Building the RNA world. Ribozymes. Curr Biol, 6: 965-7, 1996
[19] Ishizaka M, Ohshima Y, and Tani T, Isolation of active ribozymes from an RNA pool of random sequences using an anchored substrate RNA. Biochem Biophys Res Commun, 214: 403-9, 1995
[20] Mushegian AR and Koonin, EV, A minimal gene set for cellular life derived by comparison of complete bacterial genomes. Proc. Natl. Acad. Sci. USA, 93: 10268-10273.
[21] Ekland EH, Szostak JW, and Bartel DP, Structurally complex and highly active RNA ligases derived from random RNA sequences. Science, 269: 364-70, 1995
[22] Breaker RR, and Joyce GF, Emergence of a replicating species from an in vitro RNA evolution reaction.Proc Natl Acad Sci U S A, 91: 6093-7, 1994
[23] Chyba C and Sagan C, Endogenous production, exogenous delivery and impact-shock synthesis of organic molecules: an inventory for the origins of life. Nature, 355: 125-32., 1992
[24] Doudna JA, Couture S, and Szostak JW, A multisubunit ribozyme that is a catalyst of and template for complementary strand RNA synthesis. Science, 251: 1605-8, 1991
[25] Lahav N, Prebiotic co-evolution of self-replication and translation or RNA world? J Theor Biol, 151: 531-9, 1991
[26] Stadler PF, Dynamics of autocatalytic reaction networks. IV: Inhomogeneous replicator networks. Biosystems, 26: 1-19, 1991
[27] Eigen M, Gardiner W, Schuster P, and Winkler-Oswatitsch R, The origin of genetic information. Sci Am, 244: 88-92, 96, et passim, 1981
[28] Eigen M, and Schuster P, The hypercycle. A principle of natural self-organization. Springer-Verlag, isbn 3-540-09293, 1979
[29] Yockey HP, On the information content of cytochrome c. J Theor Biol, 67: 345-76, 1977
Libros Útiles
Statistics at Square One, T.D.V. Swinscow, 8th Edition Paperback, Published by Amer College of Physicians, 1983, ISBN: 0727901753
Evolution from Space, F Hoyle and Wickramasinghe, JM Dent and sons, London, 1981
Vital Dust: Life As a Cosmic Imperative, by Christian De Duve, Basic Books 1995, ISBN: 0465090451
The Major Transitions in Evolution, Maynard Smith J & Szathmary E, 1995, WH Freeman, ISBN: 0716745259
The Origins of Order: Self Organization and Selection in Evolution. By Stuart Kauffman, S. A. (1993) Oxford University Press, NY, ISBN: 0195079515.
At Home in the Universe. By Stuart Kauffman, 1995) Oxford University Press, NY.